『感染症疫学のためのデータ分析入門』
こちらの本をまとめる。お絵かきする。
- 感染と感染のメカニズムと疫学的指標
- 疫学とは、人の集団を対象とした健康や疾病に関する学問
- 人の集団の健康に関する状況や事象の分布やリスクの規定因子、影響を及ぼす規定因子(Determinants)の研究、健康問題を制御するために疫学を応用すること。
- 症例がリスク因子(感染者は保有者、スプレッダー)
- 接触様式
- 免疫と経験(ワクチン予防接種、獲得免疫)
- 緊急性
- 感染
- 微生物(細菌、ウイルス、真菌、原虫)
- 感染源に暴露されて、4通り
- キャリア、不顕性感染、顕性感染、感染なし
- その後の経過について、
- 死亡、キャリア、免疫獲得、免疫不獲得
- コッホの条件(見える、培養可能、打ったら発症、打って発症したら見える)
- 疫学三角(病因、宿主、環境)
- その特徴に合わせて、対策を打つ。
- 致命リスク、感染時致命リスク。
- インデックス・ケース(最初の特定されたケース)
- n次感染
- エピデミックは一定期間に同一の感染者が通常に比べて高い頻度で発生すること。
- それと比べて、ある地域で同一の感染症が常在的に発生することを、エンデミックという。
- エピデミックは高いとき、エンデミックは普通
- ある地域で同一の感染症が持続的に高いレベルで発生することを、ハイパーエンデミックという。
- パンデミックは、一定期間をまたいで、世界的に広範囲で同一の感染症が流行すること。
- 感染症の自然史
%matplotlib inline
import matplotlib.pyplot as plt
import numpy as np
import math
data = [1,6,53,45,34,14,10,10,3,2]
Sum = sum(data)
T = len(data)
answer = 0
for t in range(T):
return answer
answer2 = [math.log(t-gamma) for t in range(T)]
new_mu = sum(answer2)/T
cost = 10**10
candidate = (1,1,1)
for gamma in range(-100, 0, 1):
#print(cost)
if now_cost<= cost:
cost = now_cost
print(candidate)
estimated_gamma = candidate[1]
print("gamma=",estimated_gamma+12)
- 感染症と重篤度の評価
- 基本再生産数とは、集団内の全てのものが感受性を有する時に、1人の感染者が生み出す2次感染者数の平均値。
- 予防接種は、感受性をもつ人を減らす。
- 死亡率=罹患率×致命リスク
- アウトブレイク調査:能動的サーベイランス
data = [25,32,0,5]
extreme_data = [20,37,5,0]
def Factor(n):
answer = 1
if n<=1:
return 1
else:
for i in range(2,n+1):
answer *= i
return answer
def Fisher(data):
n = len(data)
child1 = Factor(data[0]+data[1])*Factor(data[0]+data[2])
child2 = Factor(data[1]+data[3])*Factor(data[2]+data[3])
mother = Factor(data[0])*Factor(data[1])*Factor(data[2])*Factor(data[3])
return child1*child2/(mother*Factor(sum(data)))
p = Fisher(data)+Fisher(extreme_data)
- 感染者割合の推定
- 感染者割合の分析
- 診断検査データを活用した推定
- 逆計算法を使用した推定
-
。ここで、aは発症者、hは新規感染者、fは潜伏期間の確率密度関数
- スケールフリーネットワーク
。kはつながりの次数、γはスケーリング変数。
-
- 捕獲再捕獲法
- 予防接種の評価
- コンパートメントモデル
- 受動的サーベイランスとそのデータ分析
- 収集、照合、解析と解釈、普及と利用
- アウトブレイクの早期検出
- 検出閾値の決定方法
- 回帰
- Stroupの方法
- 回帰
- 検出閾値の決定方法
data = [100, 200, 125]
mu = sum(data)/len(data)
def Stroup_check(new_data):
if mu - half_length <= new_data <= mu + half_length:
print("no statistical difference")
else:
print("statisical difference")
Stroup_check(240)
%matplotlib inline
import matplotlib.pyplot as plt
import numpy as np
import math
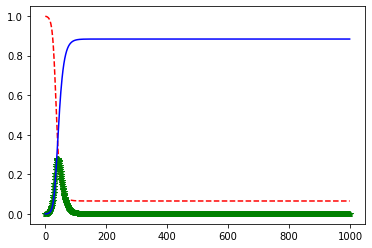
T = 1000
S = [-1]*T
I = [-1]*T
R = [-1]*T
#S = [-1]
#I = [-1]
#R = [-1]
###initial condition###
s_temp = 0.999
i_temp = 0.001
r_temp = 0
beta = 0.3
gamma = 0.1
s = [0] * T
i = [0] * T
r = [0] * T
s[0] = s_temp
i[0] = i_temp
r[0] = r_temp
for t in range(T-1):
#S = S+[-beta * S[i] * I[i]]
#I = I+[(beta * S[i] * I[i] - gamma * I[i])]
#R = R+[(gamma * I[i])]
s_temp = -beta * s_temp * i_temp + s_temp
#print(s_temp)
i_temp = beta * s_temp * i_temp - gamma * i_temp + i_temp
r_temp = gamma * i_temp + r_temp
s[t+1] = s_temp
i[t+1] = i_temp
r[t+1] = r_temp
#print(s_temp,r_temp)
import matplotlib.pyplot as plt
# プロット範囲のxを用意
time = [k for k in range(T)]
# x = array([-5, -4, -3, -2, -1, 0, 1, 2, 3, 4, 5])
# xに対応するyの値を用意
#y = x**3
# pyplot.plot(x, y)でプロット作成
plt.plot(time, s, "r--")
plt.plot(time, i, "g*")
plt.plot(time, r, "b")
# plt.show()で画面に表示
plt.show()
-
流行のモニタリング
- 報告が遅れるので、たたみ込みの式になる。
- 輸入感染例と国内感染例がある。国内感染例は全感染例からのたたみ込み。
- 2次感染のバラつきと流行発生確率
- 二次感染のバラツキ
- スーパースプレッダー
- 大規模流行の確率
- 分岐過程モデル
- 子孫の分布確率
- 絶滅確率
- 輸入感染者による流行確率
- 移動制限
- 分岐過程モデル
- 流行収束
- 二次感染のバラツキ
一旦これで止めておく。
バイバイ!