『遺伝統計学の基礎』の第2章
こちらの本に引き続き入門する。
DNA、RNA、タンパク質、形質。
- DNA2重鎖
- 染色体
- DNA分子とタンパク質からなる
- DNA分子
- 4種類の塩基が一列に並ぶ、連なる
- 配列が情報となる
- 2重であること
- AとT、GとCというペアがある。
- 片方の配列に、もう片方が対応する。
- メリット
- バックアップ
- 逆向きもすぐに取り出せる
- 複製、変異、組換え
- 起源が同じーIBD
- 1つの数値で表して扱い安くするーIBDの期待値
- 期待値で表すと、分布の情報が失われる。
- 平均、分散、モーメント、期待値
- モーメント
- cを中心としたk次のモーメント
- モーメント
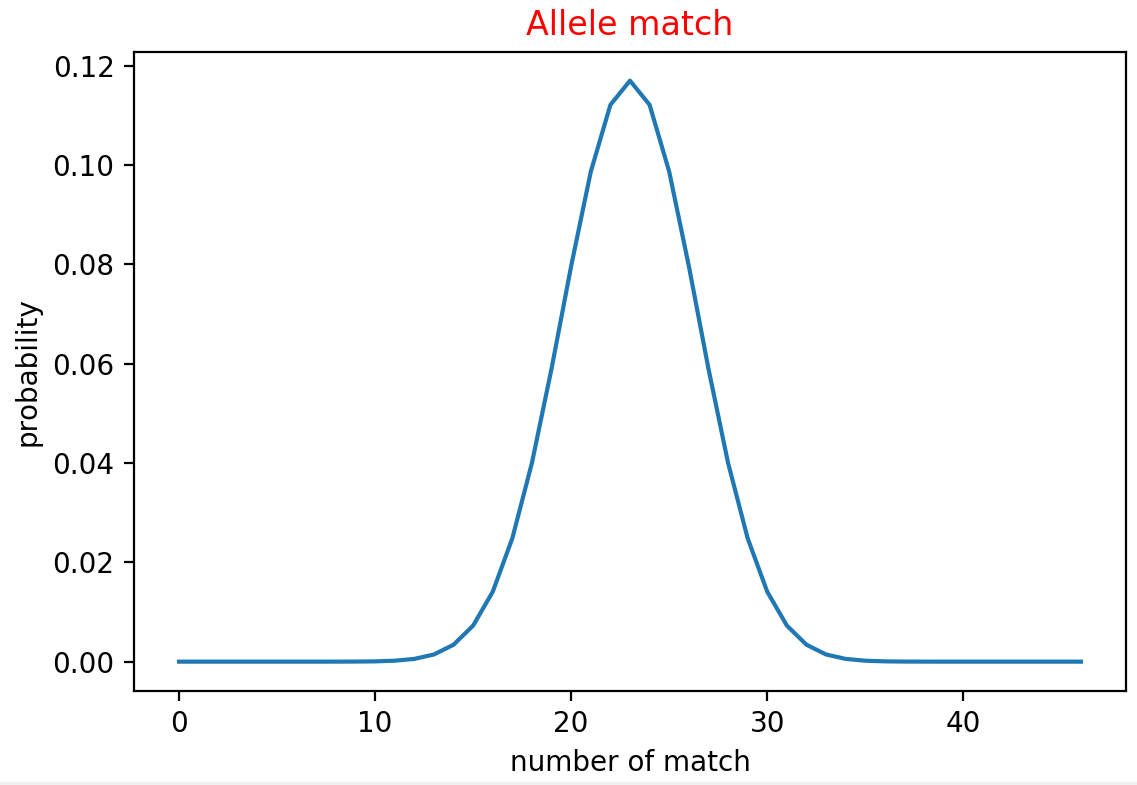
- 同胞のアレルの一致率
- 染色体の長さがバラバラ
- 染色体の長さが等しいモデルと等しくない(実際)モデル
- プロットすると、等しい方は、階段状になる。
- 教訓(気をつけること)
- データをプロットすること
- 特別な条件
- 離散的な性質
- 染色体
お絵かきする
import matplotlib.pyplot as plt
import numpy as np
import math
import random
def moment(x,p,c,k):
if len(x)==len(p):
return sum([p[i]*(x[i]-c)**k for i in range(len(p))])
else:
return None
def ave(x):
return sum(x)/len(x)
data = [1,2,3]
prob = [0.2,0.3,0.5]
mom = moment(data,prob,2,ave(data))
print(mom)
def factorial(n):
if n==0:
return 1
elif n==int(n) and n>0:
fac = 1
for i in range(1,n):
fac*=(i+1)
return fac
def combination(n,i):
return factorial(n)/(factorial(i)*factorial(n-i))
def allele(k,i):
return combination(2*k,i)/2**(2*k)
def plot_allele():
k=23
num_match=[i for i in range(2*k+1)]
prob_match=[]
for i in range(2*k+1):
prob_match.append(allele(k,i))
plt.plot(num_match, prob_match)
plt.xlabel("number of match")
plt.ylabel("probability")
plt.title("Allele match",color="red")
plt.show()
plot_allele()
-
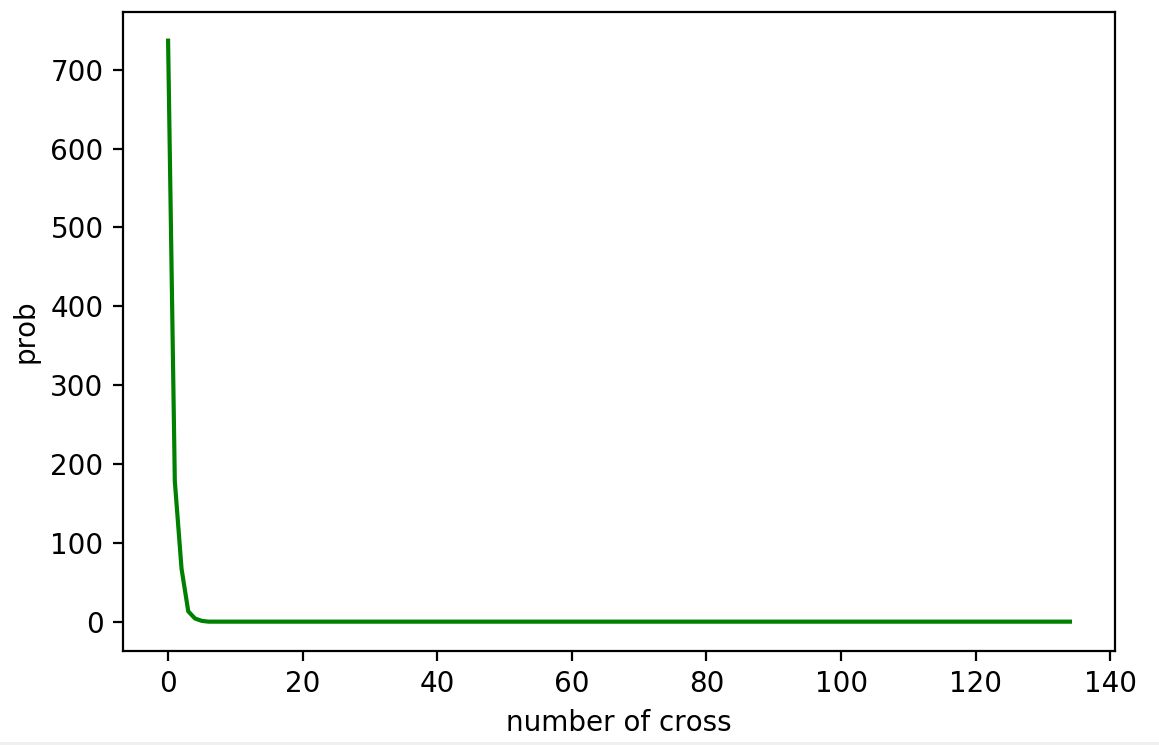
- 染色体は23対よりももっと細かくなるーー交叉、組換え:ポアッソン過程と指数分布
- 交叉が起こることは非常にレア。
- レアなことが起こる時の分布として、ポアッソン分布がある。
- 何回中××回○○が起こること。
- その××を固定して、試行回数を無限に飛ばすと、ポアソン分布
- 交叉間距離が指数分布になるらしい。
- サイコロを振って6が出ないことが続いていく様子が指数分布になっているのと同じこと??
- レアなことが起こる時の分布として、ポアッソン分布がある。
- 交叉が起こることは非常にレア。
- 染色体は23対よりももっと細かくなるーー交叉、組換え:ポアッソン過程と指数分布
データはプロットしてみなさい。という教訓があったので、お絵かきしてみる。
#initial settings
L=10000;r=0.0001;N=1000
#function settings
def poisson(l,k):
return l**k/factorial(k)*math.exp(-l)
#poisson分布を頑張って作る
#xの時の確率は分かった。
m=135
poisson_data=[0]*m
poisson_data[0]=poisson((L-1)*r,0)
for i in range(m-2):
poisson_data[i+1]=poisson_data[i]+poisson((L-1)*r,i+1)
poisson_data[-1]=1
#確率がわかった上で、そこからサンプリングするようにしたい
#二分探索で絞り込むことで、確率分布の逆関数チックなことをしている。
def binary_search(x,data):
l=0;r=len(data)
while r-l>1:
mid=(l+r)//2
if x>=data[mid]:
l=mid
else:
r=mid
return l
def main():
random_data=[random.random() for i in range(N)]
data=[0]*m
cnt=[i for i in range(m)]
for i in range(N):
num=binary_search(random_data[i],poisson_data)
data[num]=data[num]+1
plt.plot(cnt,data,color="green")
plt.xlabel("number of cross")
plt.ylabel("prob")
plt.show()
main()
- DNAからRNA、タンパク質へ
バイバイ!(←この記事の終止コドン)